
Na busca por tentar compreender por que algumas pessoas não desenvolvem covid-19, pesquisadores da USP, Unesp e do Grupo Fleury identificaram alguns genes do sistema imune humano que podem ajudar a entender esse mecanismo. Os genes candidatos são conhecidos como MICA e MICB e pertencem ao complexo MHC (complexo principal de histocompatibilidade, em português), localizado no cromossomo 6. Essas moléculas já foram descritas em estudos anteriores e estão associadas com estresse celular, como câncer e infecções.
Após comparar 86 casais em que um desenvolveu a doença e o outro não (chamados casais discordantes), os pesquisadores observaram, por modelos matemáticos (também chamados in silico), que moléculas MICA estavam aumentadas e as MICB diminuídas nos indivíduos infectados. Por outro lado, as MICB estavam aumentadas nos resistentes.

Um artigo com os resultados do estudo foi publicado no último dia 25 na plataforma MedRxiv, e ainda não foi revisado por outros cientistas. Para que o MICA e o MICB possam agir, as moléculas por eles codificadas precisam se ligar a um receptor chamado NKG2D, localizado na superfície das células natural killer (NK) e linfócitos TCD8+, principalmente.
Quando isso acontece, há ativação de células do sistema imune, com a produção de fatores inflamatórios para combater a infecção, principalmente o IFN- ϒ, e liberação de proteínas “tóxicas”, culminando com a morte das células infectadas.
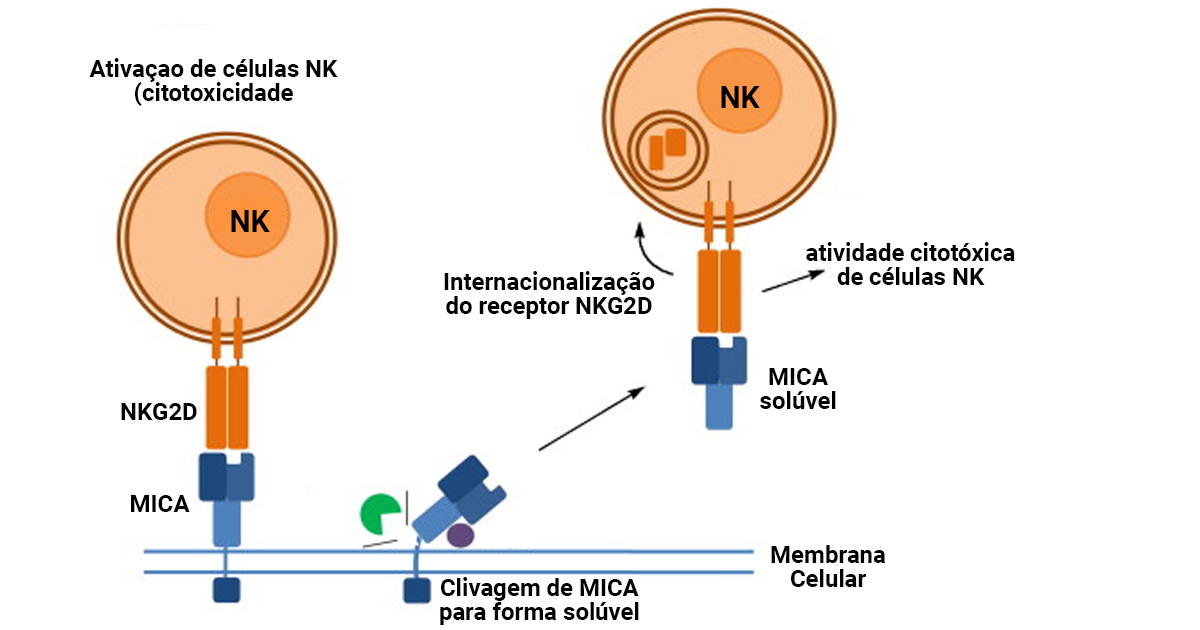
As células NK fazem parte do sistema imune inato e sua função principal é reconhecer o patógeno invasor e destrui-lo rapidamente. A hipótese dos cientistas é que as moléculas MICA aumentadas sejam clivadas (cortadas) na superfície celular infectada e passem para sua forma solúvel. Nessa mudança, elas podem “inibir” seu receptor e diminuir a atividade de células NK, facilitando o desenvolvimento da doença.
“No caso do MICB, é possível que sua expressão diminuída nos pacientes infectados possa, da mesma forma que o MICA solúvel, contribuir para uma menor ativação de células NK e linfócitos TCD8+ e uma menor resposta imune contra o vírus ”, explica ao Jornal da USP Maria Lucia Carnevale Marin, pesquisadora do Laboratório de Imunologia do Instituto do Coração (Incor) e do LIM19 do Hospital das Clínicas da Faculdade de Medicina da USP. Ela fez parte da equipe que interpretou os dados da pesquisa.
Processo de clivagem
Outro complexo estudado foi o LRC (complexo leucocitário humano), localizado em uma fração muito pequena do cromossomo 19. Após as análises, os genes LILRB1 e LILRB2 estavam cinco vezes mais expressos nos infectados do que nos não infectados. Essas são variantes que atuam como inibidores das células NK, ou seja, não protege contra a infecção.
Os casais discordantes passaram por um critério de seleção rígido para serem incluídos no estudo. Além disso, repetiram o exame de sorologia para certificar que um deles não desenvolveu anticorpos contra o vírus. Dos 100 selecionados, sete ficaram de fora da pesquisa.
“Podemos pensar, futuramente, se seria possível aumentar a expressão do MICB com a ingestão de alguma droga, por exemplo, e ajudar as células de defesa a combaterem a infecção”, afirma a professora Mayana Zatz, diretora do Centro de Estudos sobre o Genoma Humano e Células-Tronco (CEGH-CEL) da USP e coordenadora da pesquisa.
O estudo foi realizado em 2020, quando as variantes P1 e P2 do sars-cov-2 ainda não circulavam pelo País.
Mecanismos de ação

O complexo principal de histocompatibilidade (MHC) é conhecido por ser a região mais variável do genoma humano e da maioria dos vertebrados. Algumas moléculas codificadas por genes desse complexo desempenham várias funções, como a de apresentar antígenos para as células T e B citotóxicas do sistema imune, que identificam esse corpo estranho e iniciam a resposta imune contra eles.
São conhecidos como genes HLA clássicos de classe I (HLA-A, HLA-B e HLA-C) e classe II (HLA-DR e HLA-DQ). Em caso de transplantes, sua variabilidade pode se tornar um problema, pois a compatibilidade desses polimorfismos é essencial para que o novo órgão não seja rejeitado.
Já o MICA e o MICB, especificamente, atuam como sinalizadores de estresse celular. Quando uma célula é infectada, há um aumento da expressão desses genes, que interagem com receptores das células natural killer (células que apresentam uma resposta mais rápida contra infecções) e as ativam. Eventualmente, as NK conseguem destruir a célula infectada. “Por isso, são candidatos importantes a serem estudados em uma infecção pelo sars-cov-2”, explica Castelli.
O Complexo de Receptores Leucocitários (LRC) é composto de genes que, em sua maioria, codificam moléculas da família das imunoglobulinas. “Além das diferentes sequências dentro de cada um dos genes do sistema, existe mais uma complexidade: o indivíduo pode ou não tê-los”, enfatiza Castelli. O LILRB1 e LILRB2, que estavam cinco vezes mais aumentados nos indivíduos infectados, são moléculas que inibem a ação das células natural killer e também poderiam contribuir para a diminuição da resposta imune contra o vírus.
“É importante ressaltar que a maioria das doenças não está associada a um único gene”, explica Maria Lucia. “Elas fazem parte de um sistema complexo e uma ou outra molécula pode colaborar para a resistência ou suscetibilidade a uma infecção.”
O início
Mayana decidiu estudar casais discordantes quando dois conhecidos relataram um caso interessante. Ele foi infectado pelo novo coronavírus e a parceira não, apesar de compartilharem o mesmo quarto. Intrigada, ela recorreu às mídias impressa e televisiva com o objetivo de conhecer outras pessoas que passaram pela mesma situação. Segundo ela, mais de 2 mil e-mails foram recebidos. Mateus Vidigal, biólogo do CEGH-CEL e um dos autores do estudo, foi o responsável pela triagem e seleção dos voluntários. “Por questão de logística, incluímos apenas casais moradores de São Paulo”, explica o pesquisador.
Mayana relata ao Jornal da USP que os resultados preliminares surpreenderam os cientistas. “Tivemos uma diferença muito grande de gênero. O grupo dos suscetíveis era composto de 53 homens e 33 mulheres; entre os resistentes, estavam 29 homens e 57 mulheres.”
Na etapa seguinte, amostras de sangue dos voluntários passaram pela análise de exoma (conjunto de todas as regiões codificadoras – exons – do DNA humano). “Vimos que essas variantes eram comuns na população de São Paulo”, explica a geneticista. Quando isso acontece, segundo ela, logo se deduz que devem ter muitos genes atuando na mesma condição.
Para realizar os estudos, seria preciso ampliar o estudo e selecionar de 20 a 30 mil pessoas. “Decidimos focar, então, em genes do sistema imunológico”, relata. “Entramos em contato com Erick Castelli, pesquisador do Departamento de Patologia da Faculdade de Medicina da Unesp, em Botucatu, para que ele realizasse as análises dos exomas desses indivíduos.”
Trabalhando com algoritmos
Castelli estuda genes dos sistemas MHC e LRC há bastante tempo e desenvolveu um modelo matemático, junto com alunos de pós-graduação, capaz de analisá-los com mais precisão. Por causa da quantidade de variantes existentes, qualquer estratégia tradicional de investigação é incapaz de interpretar corretamente essas informações.
“Nosso método computacional utiliza algoritmos baseados em um banco de dados de sequências já conhecidas de cada gene”, explica. “Ele corrige a montagem do sequenciamento e permite que a gente consiga ver variante por variante, e também qual é a sequência que o indivíduo tem em cada cromossomo.”
Com esses dados, é possível predizer quais proteínas essas variantes podem produzir. “Por isso, conseguimos comparar resistentes e infectados em diferentes níveis”, relata.
Futuro
“O que não sabemos ainda é por que existem moléculas que foram muito ou pouco expressas”, indaga Edécio Cunha Neto, professor da Faculdade de Medicina da USP. “As MICA podem estar sendo clivadas na membrana da célula, passam a circular no sangue e as células NK não conseguem reconhecer e fazer a interação com a célula infectada.”
Ainda segundo Cunha Neto, serão necessários estudos in vitro (em laboratório) para validar os achados do modelo matemático. “Devemos medir a expressão do MICA e do MICB nas células dos indivíduos suscetíveis e resistentes e estudar a resposta das células NK frente à infecção.”


