 Mineração de cobre potencializa drenagem ácida de mina, solução aquosa formada a partir da oxidação de minerais sulfetados, como a calcopirita, com altos teores de metais pesados e acidez, que pode contaminar águas subterrâneas e solo – Foto: Cedida pelo pesquisador/Leandro Nascimento Lemos
Mineração de cobre potencializa drenagem ácida de mina, solução aquosa formada a partir da oxidação de minerais sulfetados, como a calcopirita, com altos teores de metais pesados e acidez, que pode contaminar águas subterrâneas e solo – Foto: Cedida pelo pesquisador/Leandro Nascimento Lemos
.
“Apesar dos benefícios econômicos oriundos da mineração, esta atividade potencializa a produção de drenagem ácida de mina, que é caracterizada por uma solução aquosa formada a partir da oxidação de minerais sulfetados, como por exemplo a calcopirita”, explica o biólogo Leandro Nascimento Lemos, que integrou o grupo de pesquisa. “Este material é constituído por altos teores de metais pesados e pH ácido, extremamente baixo, que pode contaminar o solo e o lençol freático, ou seja, cursos de água subterrâneos.”
De acordo com o biólogo, uma importante alternativa para controle da drenagem ácida de mina é a biorremediação. “A biorremediação é um processo que utiliza micro-organismos, especialmente do próprio ambiente, que podem auxiliar na neutralização da acidez (pH)”, descreve. “Um grupo muito usado para esse fim são as bactérias redutoras de sulfato.”
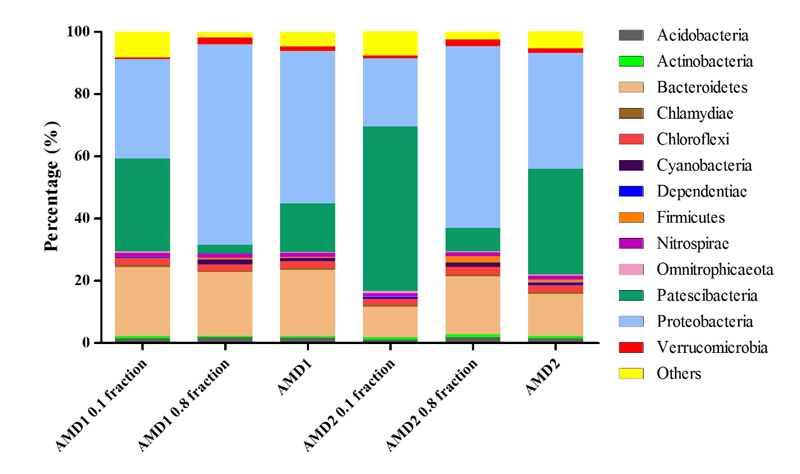
O pesquisador destaca que as interações entre os membros das comunidades microbianas presentes na drenagem ácida são fundamentais para o desempenho da biorremediação. “O objetivo da pesquisa foi investigar o metabolismo de duas novas espécies do grupo Saccharimonadia e suas interações ecológicas com outras bactérias que estão presentes em drenagem ácida de mina”, aponta. “O grande desafio foi reconstruir o genoma destes micro-organismos, tendo em vista que entre 85% e 99% deles ainda não são cultiváveis em laboratório, devido à dificuldade de simular as condições do ambiente em que vivem.”
.

.

.
Matéria escura microbiana
A dificuldade de cultivo das bactérias estudadas no trabalho faz com que sejam consideradas por especialistas em microbiologia como “microbial dark matter”, isto é, “matéria escura microbiana”. “A localização e identificação foram a partir do uso de abordagens avançadas de metagenômica (estudo de material genético recuperado diretamente de amostras ambientais) e bioinformática”, relata Nascimento Lemos. “Inicialmente foram coletadas amostras de drenagem ácida da Mina do Sossego, em Canaã dos Carajás, no Pará. “Por meio de técnicas de biologia molecular, baseadas na extração e sequenciamento massivo do DNA genômico de todas as espécies microbianas que estão nas amostras, foi possível obter uma enorme quantidade de dados.”
Por se tratar de micro-organismos que ainda não foram cultivados, os genomas dessas novas espécies foram reconstruídos a partir de fragmentos presentes em uma comunidade microbiana de alta complexidade. “Este processo é análogo à montagem de um quebra-cabeça”, conta o biólogo, “onde as peças individuais, que representam fragmentos de genomas de todas as espécies que estão na comunidade microbiana, são agrupadas com base em características comuns.”
Investigando o genoma das bactérias, os pesquisadores descobriram que elas não apresentam genes associados à biossíntese de aminoácidos, nucleotídeos, ácidos graxos e cofatores, que são moléculas fundamentais para a sobrevivência de bactérias de vida livre. “Além disso, por apresentarem um genoma muito pequeno, menor que 1 Megabase (Mbp), quando uma bactéria de solo, por exemplo, tem em média um genoma de 4.5 Mbp, foi levantada a hipótese de que elas poderiam estar vivendo em simbiose com outras bactérias”, ressalta o pesquisador.
.
Usando métodos computacionais baseados na Teoria de Redes Complexas, a pesquisa encontrou evidências de co-ocorrência e dependência metabólica com outras bactérias, tais como as do gênero Hydrotalea. “Por elas co-ocorrerem com outras bactérias que são potenciais para o processo de biorremediação, mas que ainda não foram cultivadas, no futuro elas poderiam ser aplicadas em consórcios microbianos para descontaminação de solos”, conclui Nascimento Lemos. O trabalho é descrito no artigo Genomic signatures and co‐occurrence patterns of the ultra‐small Saccharimonadia (phylum CPR/Patescibacteria) suggest a symbiotic lifestyle, publicado na revista Molecular Ecology em 25 de agosto.
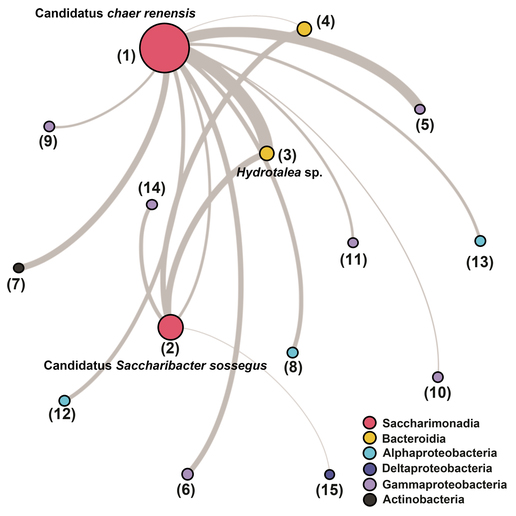
A pesquisa foi realizada por um grupo multidisciplinar, coordenado pelo professor Victor Pylro, da Universidade Federal de Lavras (UFLA), em Minas Gerais. A reconstituição do genoma das bactérias foi feita na Plataforma de Bioinformática do Instituto René Rachou, da Fundação Oswaldo Cruz (Fiocruz) de Minas Gerais. Também integraram o grupo de pesquisa Julliane Medeiros, da Universidade Federal de Juiz de Fora (UFJF), Francisco Dini-Andreote, da The Pennsylvania State University (Estados Unidos), Gabriel Fernandes, da Fiocruz Minas, Alessandro Varani, da Universidade Estadual Paulista (Unesp) e Guilherme Oliveira, do Instituto Tecnológico Vale, em Belém (Pará).
Mais informações: e-mail llemos@usp.br, com Leandro Nascimento Lemos







































